
蜂群図(ビースウォーム)の作成(R)
※2023年11月10日 ggbeeswarmについて追記。 ※2024年5月26日 boxplot()の外れ値の非表示について追記。beeswarmパッケージ
各群のデータのばらつきを、個々のデータポイントのドットプロットによって図示します。
下記では、ビースウォーム(biostatistics)を参考に作図しています。
install.packages("beeswarm", dependencies = TRUE) #未インストールの場合
library(beeswarm)
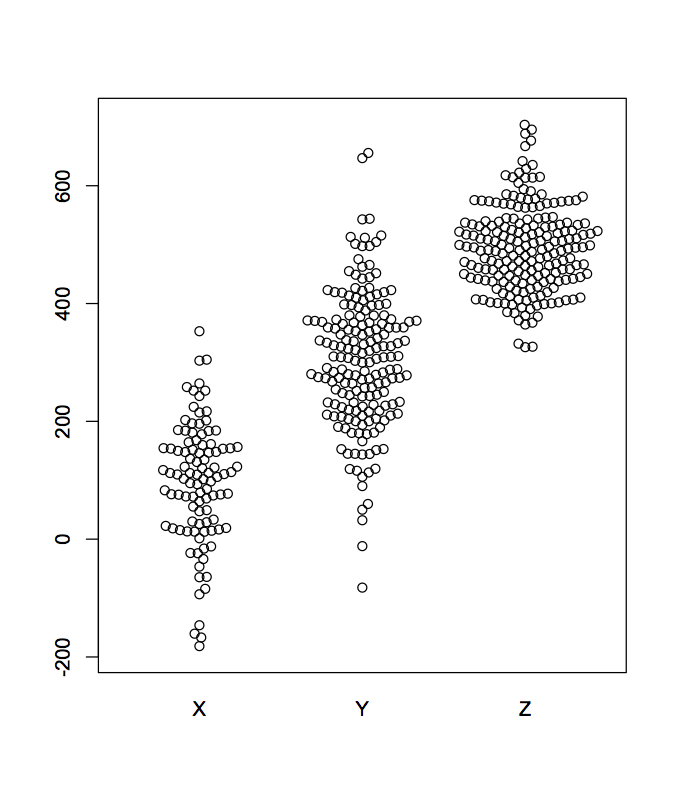
x <- rnorm(100, 100, 100) # サンプル数100、平均100、標準偏差100
y <- rnorm(180, 300, 120)
z <- rnorm(200, 500, 80)
beeswarm(list(x, y, z), labels = c("X", "Y", "Z"))
箱ひげ図と重ねます。
引数methodで、swarm、center、hex、squareから配置方法を選択できるとのこと。次の例では、デフォルトなので不要ですが、swarmを指定しています。
boxplot(x, y, z) beeswarm(list(x, y, z), method = "swarm", add = T) # add=TRUEで上書き
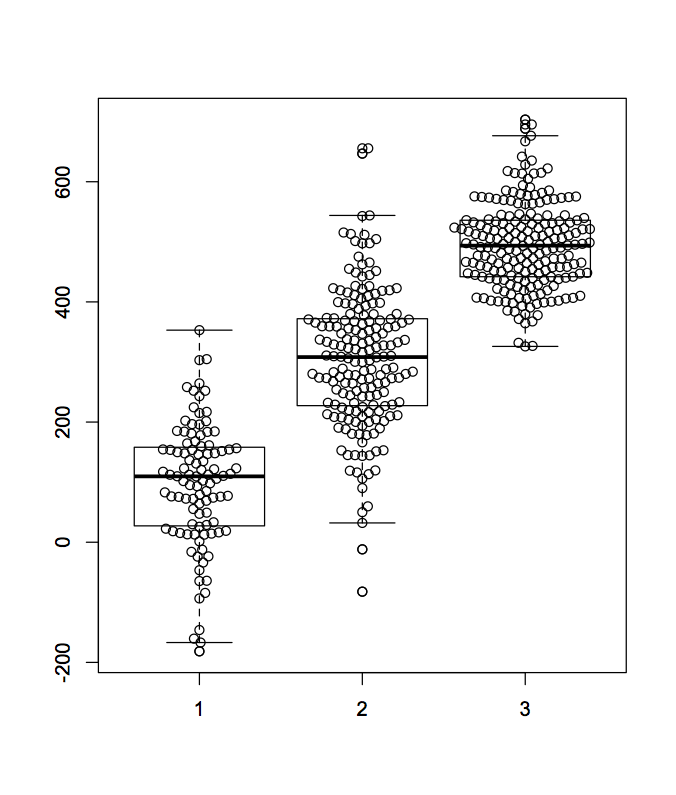
この例では、箱ひげ図と蜂群図の外れ値が微妙に位置がずれて表示されており、データポイントが増えたように見えます。
箱ひげ図の外れ値を表示させない場合は、boxplot()の引数で、outline = FALSEとしてください。
stripchart関数
さてbeeswarmの特徴は、個々のデータポイントが互いに重なり合わないようなプロットであるという点です。
stripchart()を使うと重ねてプロットしますが、引数method="jitter"にすることで、やや横に散らばらせることができます。
a <- rnorm(30,20,5) b <- rnorm(20,10,3) c <- rnorm(25,5,3) boxplot(a, b, c) stripchart(list(a, b, c), vertical = TRUE, pch = 1, method = "jitter", add = TRUE)
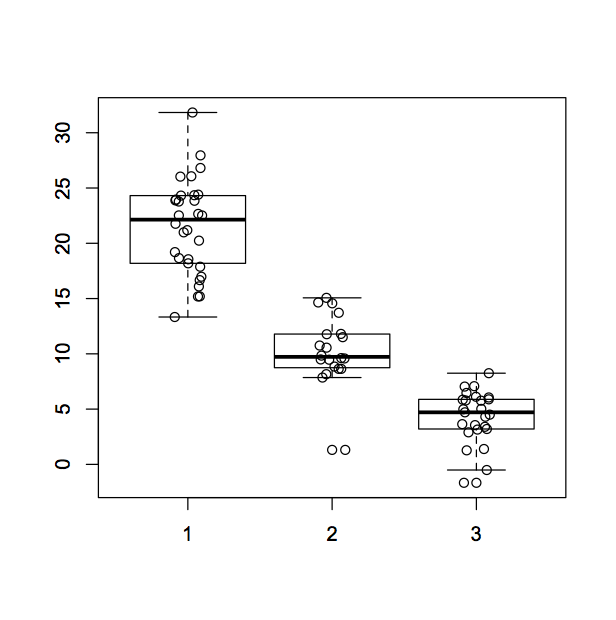
上の例では、boxplot()のマーカー(引数pch)がデフォルトで○なので、stripchart()で揃えてあります。
なお、boxplotに外れ値がある場合、stripchartのプロットと重なる可能性があります。
bxplot()の引数、outline = Fと指定すると、外れ値だけ非表示になります。
ggplot2
ggplot2ではもっと細やかに描画することができます。
ggplot2での描画では、データフレームのような整然データ(tidy data)を扱わなければなりません。
xax <- c(rep("a",length(a)),rep("b",length(b)),rep("c",length(c)))
yax <- c(a,b,c)
d <- data.frame(xax,yax)
head(d);tail(d)
# データフレームdの行冒頭と末尾の出力
xax yax
1 a 30.91408
2 a 13.06420
3 a 19.23034
4 a 19.20023
5 a 13.77287
6 a 21.32040
xax yax
115 c 2.571205
116 c 6.032043
117 c 5.499815
118 c 4.243395
119 c 8.989186
120 c 6.183650
ggplotで描画します。最初に箱ひげ図のオブジェクトを作り、次に個々のデータポイントをプロットしたオブジェクトを作ります。
次の例では、geom_jitter()で個々のデータを描画します。最後にplot()で、箱ひげ図と点プロットを重ねた図を描画します。
install.packages("ggplot2", dependencies=TRUE) #未インストールの場合
library(ggplot2)
g <- ggplot(d,aes(x=xax,y=yax,colour=xax))
g <- g + geom_boxplot(outlier.shape = NA) #外れ値のプロットを省く
g <- g + geom_jitter()
plot(g)
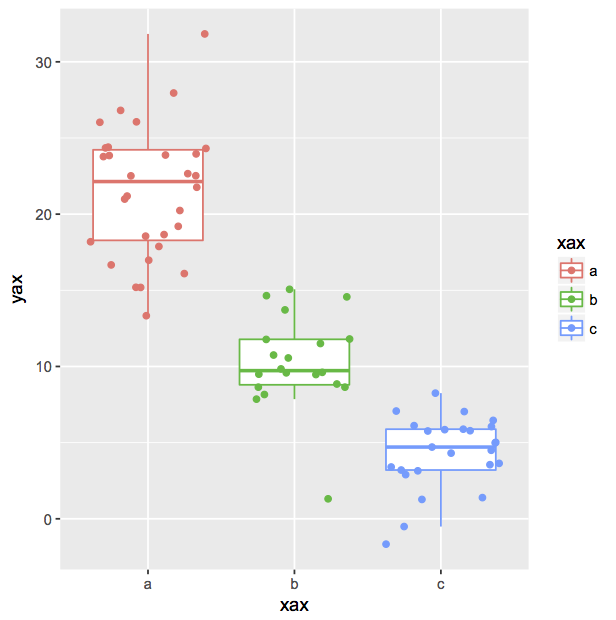
点プロットをgeom_point()で描画することもできます。
g <- ggplot(d,aes(x=xax,y=yax,colour=xax)) g <- g + geom_boxplot(outlier.shape = NA) g <- g + geom_point(position = position_jitter(width=0.05)) plot(g)
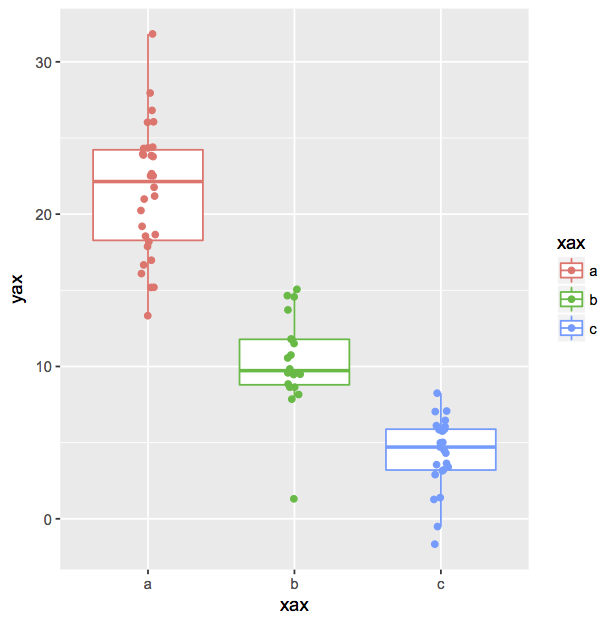
さてここまで紹介してきたプロットでは、重なりの有無の違いはあるものの、ドットを横軸に対しランダムに配置しています。
したがって、配置はプロットするごとにランダムに変化します。
library(grid) # gridを使ってggplotのオブジェクトを描画
grid.newpage()
l <- grid.layout(nrow=2, ncol=5) # 描画領域を2行5列に分割
grid.show.layout(l) #レイアウトの確認
v <- viewport(layout=l)
pushViewport(v)
d2 <- data.frame(x=rep("a",length(a)), y=a)
n <- 0
col <- 0
for(i in 1:10){
if(i == 6){ n <- n + 1; col <- 0 }
col<-col+1
g <- ggplot(d2, aes(x=x, y=y)) + geom_boxplot(outlier.shape = NA) +
geom_boxplot(outlier.shape = NA) + geom_point(position=position_jitter(width=0.5))
print(g,vp=viewport(layout.pos.row=n+1, layout.pos.col=col))
}
popViewport()
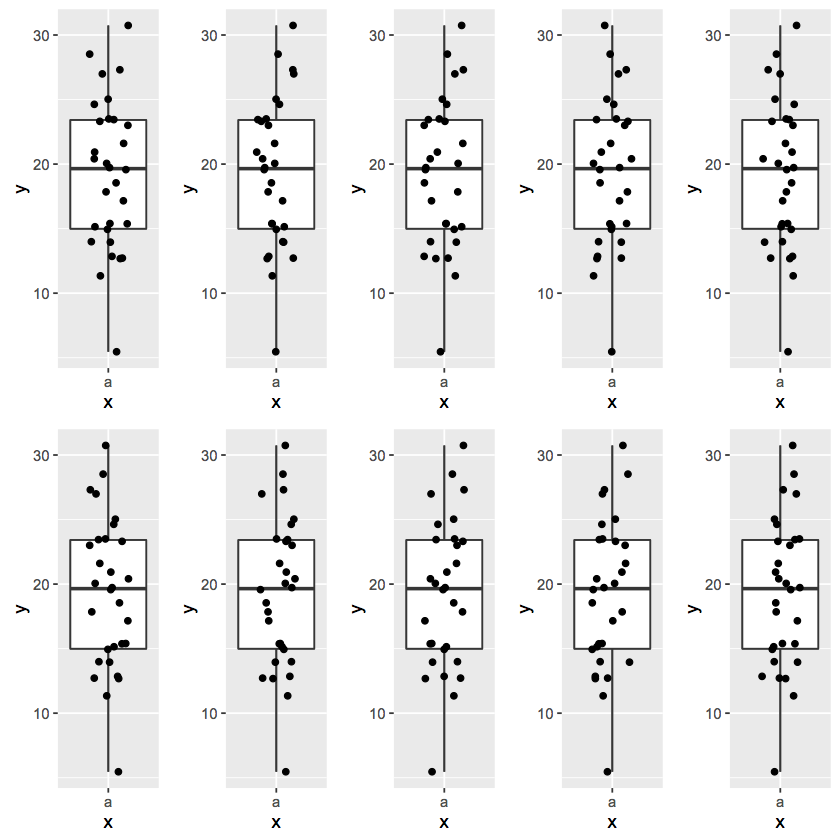
プロットのx軸座標がその都度変化していることがわかります。
なおggplot2のver.2.3では、position_jitter()の引数にseedを指定することが出来るようになり、以前の描画でのドットの配置を再現できるようになるそうです。
ggbeeswarm
上の例では各データポイントが水平にランダムに配置(ジッター)されていますが、データにタイがあると、垂直方向の重なりが増え、見づらくなります。
そこで、ggplot2では、ggbeeswarm::geom_beeswarm()を使うことでも重なりのない描画ができます。
library(ggplot2)
library(ggbeeswarm) #まだの場合はパッケージをインストール
library(ggsci) #ggplot用の色スタイルのライブラリ
#サンプルデータセットのInsectSprays使用
g<-ggplot(data=InsectSprays, aes(x=spray, y=count, colour=spray))+
geom_boxplot(outlier.shape=NA)+
geom_beeswarm(size=1,cex=2)+
scale_colour_nejm()+guides(colour="none",fill="none")
ggsave('InsectSprays_ggbeeswarm_cex2.png',g,width=4,height=3)
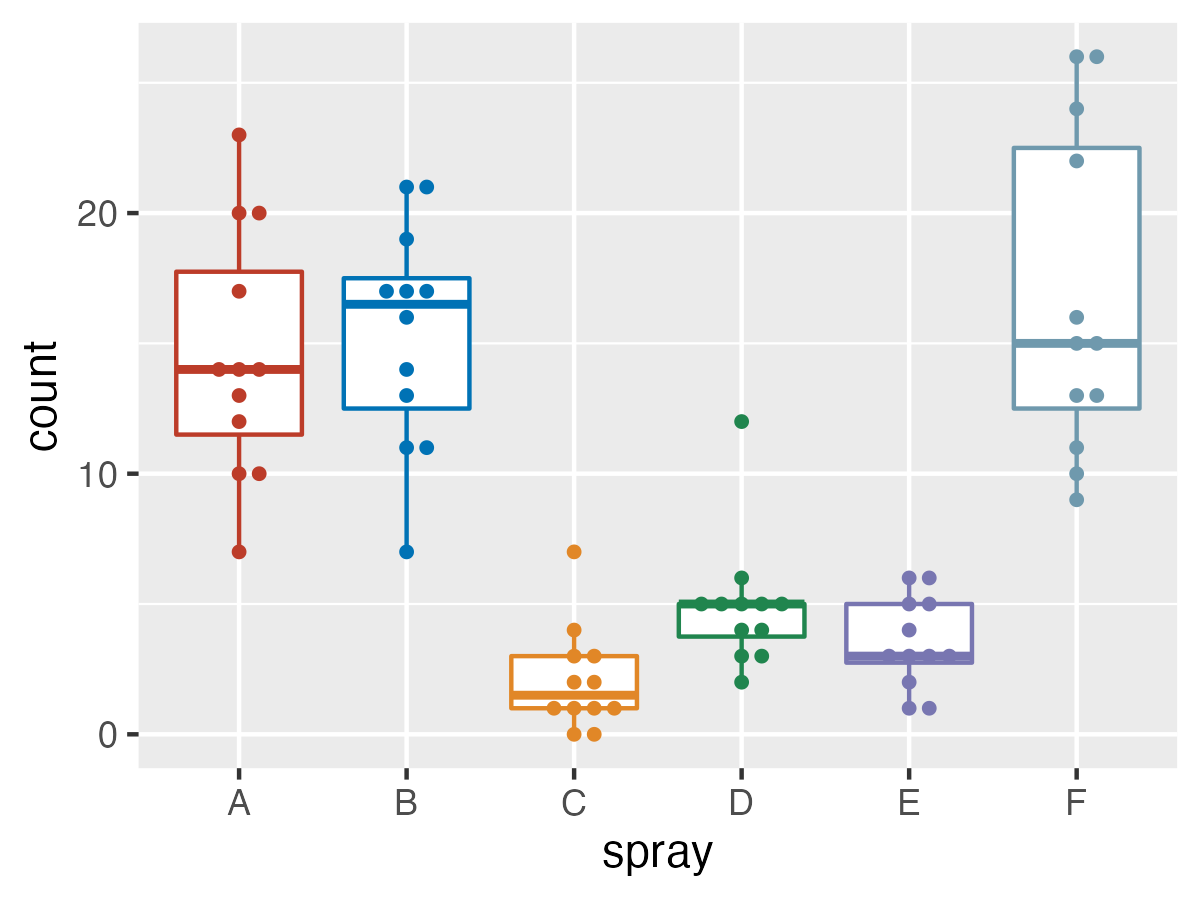 geom_beeswarm(cex=2)の場合。
geom_beeswarm(cex=2)の場合。
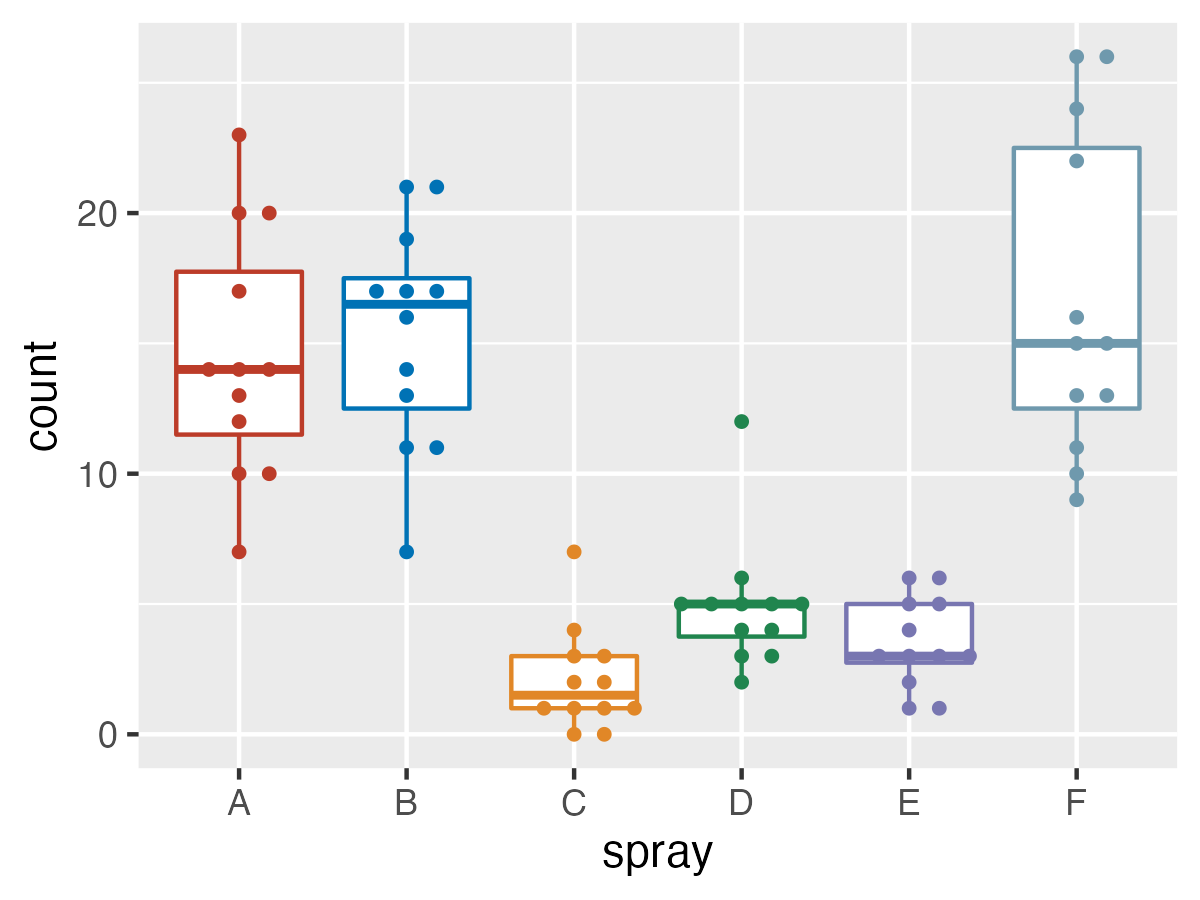 geom_beeswarm(cex=3)の場合。
geom_beeswarm(cex=3)の場合。